15. IDENTIFICANDO BASTONETES GRAM-NEGATIVOS
Como referenciar esta página: Nanes-Lima, Z.; Vieira, R.C.R., Silva, L.S. Identificando Bastonestes Gram-negativos. Detalhes Microbiológicos, Campina Grande, 09 nov. 2025. Disponível em: https://microimunoliga.blogspot.com/2016/09/identificando-bastonetes-gram-negativos.html#more 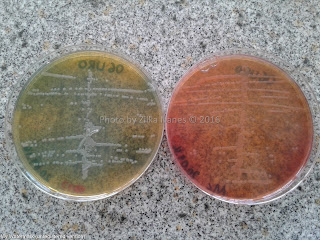 |
| Salmonella spp. em ágar CLED e ágar MacConkey após 24 horas de incubação à 36ºC. Nos dois meios de cultura observamos ausência de fermentação da lactose. Fonte: Nanes-Lima (Arquivo pessoal) |
(Microbiologia Básica / Profa . Zilka Nanes Lima)
Página atualizada em 09/11/2025
Creative Commons(by-nc-nd) ZILKA NANES LIMA(2025).
Atribuição-NãoComercial-SemDerivações.
IDENTIFICANDO BASTONETES GRAM-NEGATIVOS
(Enterobactérias e Pseudomonas aeruginosa)
ATIVIDADE PRÁTICA: Número da amostra ( )
1. Descreva
a morfologia das colônias em ágar CLED, ágar MacConkey e ágar EMB:
1. a) Descreva as características morfológicas das colônias bacterianas isoladas em ágar CLED.
1. a) Descreva as características morfológicas das colônias bacterianas isoladas em ágar CLED.
Tamanho: ________________________ Cor: _______________________________
Borda ou forma:___________________ Densidade: _____________________________
Borda ou forma:___________________ Densidade: _____________________________
Elevação:_________________________ Consistência: ___________________________
Odor: ___________________________ Hemólise (em ágar Sangue):________________
1. b) Descreva as
características morfológicas das colônias bacterianas isoladas em ágar MacConkey
e ágar EMB.
Tamanho: ________________________ Cor: ________________________________
Borda ou
forma:___________________ Densidade: ___________________________
Elevação:_________________________ Consistência: _________________________
Odor: ___________________________
1.c) Observando estas características morfológicas, qual(is) é
(são) a(s) sua(s) suspeita(s) ?
__________________________________________________________________________________________________________________________________________________________________
2.Realize o Gram de uma ou poucas
colônias deste micro-organismo, depois descreva observando à microscopia
(x1.000 - imersão) a forma, o arranjo (se houver) e a afinidade pelo corante.
[Obs: Se bastonetes relatar se são curtos (alguns aparentando cocobacilos),
médios ou longos.]:
__________________________________________________________________________________________________________________________________________________________________
3. Identifique o(s) micro-organismo(s) isolado através de testes bioquímicos tradicionais, baseando-se também nas características morfológicas das colônias em ágar MacConkey. Utilize as tabelas de +/- de identificação de enterobactérias. De acordo com a sua suspeita direcione a análise para a(s) tribo(s). Caso não consiga concluir a identificação apenas pela tabela anterior, prove a identificação através do desempate das probabilidades na tabela de percentagens. (Em anexo)
3. Identifique o(s) micro-organismo(s) isolado através de testes bioquímicos tradicionais, baseando-se também nas características morfológicas das colônias em ágar MacConkey. Utilize as tabelas de +/- de identificação de enterobactérias. De acordo com a sua suspeita direcione a análise para a(s) tribo(s). Caso não consiga concluir a identificação apenas pela tabela anterior, prove a identificação através do desempate das probabilidades na tabela de percentagens. (Em anexo)
Os meios de cultura
que serão utilizados serão: Citrato / TSI / LIA / FEN / Hid. da uréia / SIM /
MIO / Caldo Malonato / 2 tubos de caldo MR-VP
Depois de identificar a
bactéria coloque ao lado de cada teste a percentagem de positividade da
bactéria que você identificou:
Catalase ( + ) 100%
Lisina descarboxilase:( ) ____
Oxidase ( - ) 0 %
Ornitina: ( ) ____
Indol: ( ) ____
Malonato: ( ) ____
VM: ( )
____
Motilidade/ 36oC : (
) ____
VP: ( )
____
D-glicose (ácido): ( ) ____
Citrato: ( ) ____
D-glicose (gás/CO2): (
) ____
Sulfeto de hidrogênio (H2S):
( ) ____ Lactose: ( ) ____
Hidrólise da uréia: ( ) ____ Sacarose: ( ) ____
Fenilalanina: ( ) ____ DNAse/ 25oC
: ( ) ____
Teste
Complementar: Lisina desaminase: (
) Não precisa pesquisar percentagem de positividade
Bactéria :
____________________________________________
4. Use o sistema numérico
para tentar identificar as enterobactérias (Não identifica Bastonetes Gram-negativos não fermentadores de glicose):
(EPM/ Mili/ Citrato + lactose
da placa de ágar MacConkey):
Lactose = Uréia= Indol=
Gás= LTD= Lisina=
H2S= Motilidade= Citrato=
____________________________________________________________
Número encontrado:________
A bactéria foi identificada
como:_________________________________________
(Nem sempre o sistema numérico identifica a bactéria de maneira
correta, por isso confirme através dos testes bioquímicos tradicionais o
resultado obtido acima)
SOBRE O SISTEMA
NUMÉRICO PARA IDENTIFICAÇÃO DE ENTEROBACTÉRIAS:
As
8 reações bioquímicas (lactose, gás, H2S, urease, desaminação do
L-triptofano (fenilalanina), motilidade, indol, lisina descarboxilase e citrato) somadas ao da fermentação da
lactose na placa de isolamento foram agrupadas em 3 conjuntos de provas. A soma
de cada um destes conjuntos produz um algarismo, de modo que o resultado produz
um número de 3 dígitos, que identifica uma espécie bacteriana. Os 3 conjuntos
de provas e seus valores são:
PRIMEIRO ALGARISMO (A) SEGUNDO ALGARISMO (B) TERCEIRO ALGARISMO (C)
Lactose (4) Urease (4) Indol (4)
Gás (2) LTD (2) Lisina (2)
H2S (1) MOT (1) Citrato (1)
Observações:
1)
Quando a reação da cepa investigada é positiva (+), colocamos o número
correspondente.
Exemplos:
Urease + = 4, Lisina + = 2. Se a reação é negativa (-), colocamos para a mesma
o valor 0(zero).
2)
Em seguida somamos os valores obtidos em cada série. O valor de A indica o
primeiro algarismo, o valor de B o segundo e o valor de C o terceiro algarismo.
Por
exemplo, uma cepa que apresente o seguinte resultado bioquímico:
Lactose -
0 Urease + 4 Indol -
0
Gás - 0 LTD + 2
Lisina -
0
H2S + 1 MOT +
1 Citrato + 1
______________________________________________________
1 7 1
terá o
número 171 que corresponde na relação à bactéria Proteus mirabilis.
*
Conferir na tabela de percentagem de enterobactérias (elaborada pela Professora
Zilka usando o livro de Koneman como referência) se todos os resultados dos
testes bioquímicos baterm com o encontrado pelo Sistema Numérico. (INFORMAÇÃO
PESSOAL)
4)
Quando no número encontrado existir mais de uma espécie bacteriana, deverão ser
realizadas provas bioquímicas complementares.
5)
Também deverão ser usadas provas complementares quando o resultado obtido
corresponde a um número não existente na tabela.
NÚMERO ESPÉCIE BACTERIANA
000 Shigella sonnei - Yersinia pestis - Shigella spp
004 Shigella spp - Yersinia enterocolitica - Escherichia coli
006 Escherichia coli
011 Citrobacter
freundii
012 Hafnia alvei
013 Serratia marcescens - Hafnia alvei
014 Escherichia coli
015 Citrobacter diversus
016 Escherichia coli
035 Providencia stuartii - Providencia alcalifaciens
040 Yersinia pseudotuberculosis - Yersinia enterocolitica
044 Yersinia enterocolitica
051 Citrobacter freundii
053 Serratia marcescens
055 Citrobacter diversus
070 Proteus penneri
074 Morganella morganii subsp.morganii
075 Providencia stuartii - Providencia rettgerii
111 Citrobacter freundii
112 Salmonella Typhi
113 Salmonella spp.
151 Citrobacter freundii
170 Proteus mirabilis - Proteus penneri
171 Proteus mirabilis
174 Proteus vulgaris
175 Proteus vulgaris
202 Hafnia alvei
204 Escherichia coli
206 Escherichia coli
210 Salmonella Paratyphi A - Serratia liquefaciens
211 Citrobacter freundii - Serratia liquefaciens
212 Hafnia alvei - Salmonella Choleraesuis - Serratia liquefaciens
213 Serratia marcescens - Salmonella Choleraesuis - Serratia liquefaciens
214 Escherichia
coli
215 Citrobacter diversus
216 Escherichia coli
235 Providencia alcalifaciens
251 Citrobacter freundii
253 Serratia marcescens
255 Citrobacter diversus
270 Proteus penneri
274 Morganella morganii
310 Salmonella Paratyphi A
311 Citrobacter freundii
312 Salmonella Choleraesuis
313 Salmonella Choleraesuis - Salmonella spp
316 Edwardsiella tarda
351 Citrobacter freundii
370 Proteus mirabilis - Proteus penneri
371 Proteus mirabilis
374 Proteus vulgaris
375 Proteus vulgaris
404 Escherichia coli
406 Escherichia coli
410 Serratia liquefaciens
411 Citrobacter freundii - Enterobacter cloacae - Serratia spp.
412 Serratia liquefaciens
413 Serratia spp.
414 Escherichia coli
415 Citrobacter diversus
416 Escherichia
coli
417 Serratia odorifera
443 Klebsiella pneumoniae subsp. pneumoniae
447 Klebsiella oxytoca
451 Citrobacter freundii - Enterobacter cloacae
455 Citrobacter diversus
511 Citrobacter freundii
551 Citrobacter freundii
604 Escherichia coli
606 Escherichia coli
607 Klebsiella oxytoca
610 Serratia liquefaciens
611 Citrobacter freundii - Enterobacter cloacae - Serratia sp
612 Serratia liquefaciens
613 Klebsiella aerogenes (Enterobacter aerogenes) - Serratia spp.
614 Escherichia coli
615 Citrobacter diversus
616 Escherichia coli
643 Klebsiella pneumoniae subsp.pneumoniae
647 Klebsiella oxytoca
651 Citrobacter freundii - Enterobacter cloacae
655 Citrobacter diversus
711 Citrobacter freundii
751 Citrobacter freundii
5. Caso a bactéria tenha sido
identificada como Pseudomonas aeruginosa
(ou outro bastonete Gram-negativo que não seja enterobactéria), anote quais
foram as características observadas para chegar a esta conclusão, além dos resultados
que queira complementar. [*É que se
espera]
- Lactose em ágar MacConkey/ ágar EMB:(
___________) *Negativa
- Crescem em ágar MC à 42ºC (_____________)
* Crescem
- Hemólise em ágar Sangue
(_______________) *Beta-hemolisina
- Citocromo-oxidase (_____________) *Positiva – reação rápida em até 10 segundos
- Fermentação da glicose em TSI (____________)
*Não fermentador da glicose
- Motilidade no SIM (_______) *Presente
(bactéria uniflagelar)
- Lisina descarboxilase (____________) *Negativa
- Sensibilidade à Polimixina B (halo ≥12
mm = Sens.) (_________) *Sensível
- Odor característico (____________) *de aminas (uva ou “sabonete de rodoviária)
- Produção de pigmento
(__________________) *Em geral pigmento verde (piocianina)
- PYR (___________) * Variável – Prova
complementar
- Esculina (___________) *Negativa – Prova
complementar
Referências:
- Oplustil, C.P.; Zoccoli, C.M.; Tobouti, N.R.; Sinto, S.I. Procedimentos Básicos em Microbiologia Clínica, 3ª Edição. Ed. Sarvier, São Paulo, SP, 2010.
- Enterokit B (Probac do Brasil/ Declaro que não tenho conflito de interesses). ENTEROKIT B (PROBAC) Acesso em 09 de novembro de 2025.
Creative Commons(by-nc-nd) ZILKA NANES LIMA(2025). Atribuição-NãoComercial-SemDerivações.
É permitido compartilhar o conteúdo com crédito às autoras, sem uso comercial e sem modificações.
Creative Commons(by-nc-nd) ZILKA NANES LIMA(2025).
Atribuição-NãoComercial-SemDerivações.
FOTOS DAS ATIVIDADES PRÁTICAS:
IDENTIFICAÇÃO DE BASTONETES GRAM-NEGATIVOS
DATA DA AULA PRÁTICA: 29 de Setembro de 2016.
Declaro que não tenho conflito de interesses (Professora Zilka Nanes Lima)
Bactéria 01 - Família Pseudomonadaceae
Pseudomonas aeruginosa (ATCC 27853) - Não é uma enterobactéria
(Bastonetes Gram-negativos, Não fermentador de glicose, citocromo oxidase positiva)
Pseudomonas aeruginosa (ATCC 27853) - Não é uma enterobactéria
(Bastonetes Gram-negativos, Não fermentador de glicose, citocromo oxidase positiva)
Bactéria 02 - Família Enterobacteriaceae
No sistema numérico - 616 - Escherichia coli (ATCC 25922)
(Bastonetes Gram-negativos, fermentador de glicose, citocromo oxidase negativa)
 |
| Bactéria 02: Escherichia coli |
 |
| Bactéria 02: Escherichia coli |
 |
| Bactéria 02: Escherichia coli |
 |
| Bactéria 02: Escherichia coli |
 |
| Bactéria 02: Escherichia coli |
Bactéria 03 - Família Enterobacteriaceae
No sistema numérico-643- Klebsiella pneumoniae subsp. pneumoniae (ATCC 700 603)
(Bastonetes Gram-negativos, fermentador de glicose, citocromo oxidase negativa)
 |
| Bactéria: Klebsiella pneumoniae subsp. pneumoniae |
 |
| Bactéria: Klebsiella pneumoniae subsp. pneumoniae |
 |
| Bactéria: Klebsiella pneumoniae subsp. pneumoniae |
 |
| Bactéria: Klebsiella pneumoniae subsp. pneumoniae |
 |
| Bactéria: Klebsiella pneumoniae subsp. pneumoniae |
 |
| Bactéria: Klebsiella pneumoniae subsp. pneumoniae |
Bactéria 04 - Família Enterobacteriaceae
No sistema numérico - 370 - Proteus mirabilis - Proteus penneri (isolado clínico de urina)
(Bastonetes Gram-negativos, fermentador de glicose, citocromo oxidase negativa)
 |
| Bactéria: Proteus mirabilis |
 |
| Bactéria: Proteus mirabilis |
 |
| Bactéria: Proteus mirabilis |
 |
| Bactéria: Proteus mirabilis |
 |
| Bactéria: Proteus mirabilis |
Bactéria 05 - Família Enterobacteriaceae
Serratia marcescens
No sistema numérico - 213 - Serratia marcescens - Salmonella Choleraesuis - Serratia liquefaciens
(Bastonetes Gram-negativos, fermentador de glicose, citocromo oxidase negativa)
 |
| Bactéria: Serratia marcescens |
 |
| Bactéria: Serratia marcescens |
 |
| Bactéria: Serratia marcescens |
 |
| Bactéria: Serratia marcescens |
 |
| Bactéria: Serratia marcescens |
Bactéria 06 - Família Enterobacteriaceae
No sistema numérico - 374 - Proteus vulgaris (isolado clínico de urina)
O teste da Ornitina-descarboxilase é Positiva sendo a bactéria 06 identificada como Morganella morganiii subsp. morganii
(Bastonetes Gram-negativos, fermentador de glicose, citocromo oxidase negativa)
 |
| Bactéria: Morganella morganii Pelo sistema numérico; Proteus vulgaris |
 |
| Bactéria: Morganella morganii Pelo sistema numérico; Proteus vulgaris |
 |
| Bactéria: Morganella morganii Pelo sistema numérico; Proteus vulgaris |
 |
| Bactéria: Morganella morganii Pelo sistema numérico; Proteus vulgaris |
 |
| Bactéria: Morganella morganii Pelo sistema numérico; Proteus vulgaris |
Texto do e-mail enviado para turma 2016.1 de Microbiologia Clínica no dia 30/09/16.
Sobre a Atividade Prática de Identificação de Bastonetes Gram-negativos e Sistema Numérico para Identificação de Enterobactérias, fecho a explicação na aula teórica da próxima quinta-feira (06/10/2016).
Os números que vocês encontraram utilizando o Sistema Numérico, e me mostraram o resultado escrito, ontem (30/09/16) durante a aula são os que seguem:
Bactéria 03 - 643 - Klebsiella pneumoniae subsp. pneumoniae
Bactéria 04 - 371 - Proteus mirabilis Errata: 370 - Proteus mirabilis - Proteus penneri
Bactéria 05 - 213 - Serratia marcescens - Salmonella Choleraesuis - Serratia liquefaciens (Pesquisar qual destas bactérias produz pigmento rosa.)
Bactéria 06 - 374 - Proteus vulgaris
Se o Sistema Numérico aponta somente um gênero-espécie, a probabilidade de ser essa enterobactéria é alta (bactérias 03, 04 e 06).
Números que indicam mais de um gênero-espécie, precisamos encontrar características extras para concluir a identificação (como é o caso da bactéria 05).
O Sistema Numérico funciona bem, mas infelizmente tem vezes que existe falha pois estamos trabalhando com seres vivos metabolicamente ativos que não necessariamente expressam concomitantemene todas as características que lhe são peculiares. Nessas bactérias que utilizei na aula prática, ele identifica a bactéria 06 com Proteus vulgaris, quando conferimos os outros testes notamos que Proteus vulgaris não tem Ornitina-descarboxilase positiva; a identificação seria Morganella morganii subsp. morganii
O Sistema Numérico acerta a identificação das bactérias 03 e 04.
Bom. Na próxima aula (06/10/16) acaberemos de preencher (e entender) esta atividade prática.
Abraços.
Profª. Zilka Nanes Lima
Creative Commons(by-nc-nd) ZILKA NANES LIMA(2025). Atribuição-NãoComercial-SemDerivações.
É permitido compartilhar o conteúdo com crédito às autoras, sem uso comercial e sem modificações.
Creative Commons(by-nc-nd) ZILKA NANES LIMA(2025).
Atribuição-NãoComercial-SemDerivações.







como faço pra achar as outras bactérias ? só vai até a 06, queria achar pelo menos a 012 ( hafnia )????????
ResponderExcluirO1 URO, 02 URO, 03 URO, 04 URO, 05 URO e 06 URO são números aleatórios que coloquei para os alunos identificarem as bactérias. Já 012 é o resultado encontrado quando usamos a regra do Sistema Numérico para Hafnia alvei.
Excluir